Analyse und Überwachung von Überschwemmungen mit Python und Sentinel-2 Daten auf EO-Lab
In diesem Beispiel werden Sentinel-2 Daten der von der Flutkatastrophe in Mittelgriechenland im September 2023 betroffenen Gebiete auswerten.
Voraussetzungen
Nr. 1 Konto
Sie benötigen ein EO-Lab Konto mit Zugriff auf die Horizon-Schnittstelle: https://cloud.fra1-1.cloudferro.com/auth/login/?next=/.
Nr. 2 Installation von Jupyter Notebook
Der Code in diesem Artikel läuft auf Jupyter Notebook. Sie können Jupyter Notebook auf der Plattform Ihrer Wahl installieren, indem Sie der offiziellen Jupyter Notebook-Installationsseite folgen.
Eine besondere Art der Installation (die nicht erforderlich ist, um diesen Artikel zu folgen), ist die Installation auf einem Kubernetes-Cluster. Dieser Artikel beschreibt das entsprechende Vorgehen:
/kubernetes/Installing-JupyterHub-on-Magnum-Kubernetes-cluster-in-EO-Lab-cloud.
Nr. 3 Daten aus dem CREODIAS-Datenbrowser herunterladen
Die Satellitendaten für die Hochwasseranalyse werden von der CREODIAS-Plattform bezogen, die lange Zeitreihen von Satellitendaten anbietet. Diese Produkte stammen von unterschiedliche EO Sensoren, wie z. B. Sentinel oder Landsat. Die Nutzer von CREODIAS können nach Produkten suchen und sie nach Zeitspanne, Ort oder Produktname filtern. Das Repository enthält Daten aus verschiedenen Zeiträumen rund um die Welt.
Die folgenden Artikel erklären die Grundlagen der Arbeit mit Data Explorer:
Anmeldung beim Data Explorer auf EO-Lab FRA1-1 Cloud.
Herunterladen eines einzelnen Produkts mit Data Explorer auf EO-Lab FRA1-1.
Verarbeitung von Produkten mit Data Explorer auf EO-Lab FRA1-1.
Schritt 1 Auswahl der geeigneten Bilder
Der erste Schritt ist die Auswahl der geeigneten Sensoren und das Herunterladen der entsprechenden Datenprodukte. Der hier vorgestellte Ansatz basiert auf der Analyse des MNDWI (Modified Normalized Differencial Water Index). Dazu benötigen wir Satellitenbilder von Zeiträumen vor und während der FLut, die im optischen Bereich des Spektrums arbeiten, und
Wellenlängen zwischen 490 und 610 Nanometern (GRÜN, z.B. Sentinel-2 BAND 3), sowie
den kurzwelligen Infrarot-Bereich (SWIR, z.B. Sentinel-2 BAND 11) abdecken.
Es wird empfohlen, atmosphärisch korrigierte (Level 2) Datenprodukte zur Analyse zu verwenden. Außerdem werden wir den Scene Classification Layer (SCL) verwenden, um die Wolkenbedeckung zu maskieren, die das Endergebnis der Analyse beeinflussen kann. Nach dem Herunterladen öffnen wir alle Bilder in einem Python-Notebook.
#import libaries
import rasterio
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.colors import LinearSegmentedColormap
input_band_3_b = 'band_3_before_flood.jp2'
input_band_11_b = 'band_11_before_flood.jp2'
input_scl_b = 'scl_image_before_flood.jp2'
input_band_3_f = 'band_3_during_flood.jp2'
input_band_11_f = 'band_11_during_flood.jp2'
input_scl_f = 'scl_image_during_flood.jp2'
Öffnen der Rasterdaten
def open(raster):
with rasterio.open(raster) as src:
raster = src.read(1)
return raster
#Get CRS and transformation data
with rasterio.open(input_scl_f) as src:
crs = src.crs
transform = src.transform
band_green_b= open(input_band_3_b)
band_swir_b = open(input_band_11_b)
scl_b = open(input_scl_b)
band_green_f = open(input_band_3_f)
band_swir_f = open(input_band_11_f)
scl_f = open(input_scl_f)
Schritt 2 Berechnung des MNDWI-Index für die ausgewählten Datensätze
Pixelwerte in Rasterbildern normalisieren
Für unseren Zweck ist es ausreichend, zwischen 0 und 255 normalisierte Pixelwerte zu verwenden. Anschließend berechnen wir den MNDWI-Indexwert anhand der Formel:
MNDWI = (GREEN – SWIR) / (GREEN + SWIR)
def normalize(band_green, band_swir):
#Calculate maximum values for each raster file
max_value = band_green.max()
max_value = band_swir.max()
#Normalize raster data to 0-255 scale
scaled_band_green = (band_green / max_value) * 255
scaled_band_swir = (band_swir/ max_value) * 255
return scaled_band_green, scaled_band_swir
#Calculate MNDWI value for given images
def mndwi(scaled_band_green, scaled_band_swir):
mndwi = (scaled_band_green - scaled_band_swir) / (scaled_band_green + scaled_band_swir)
return mndwi
band_green_b, band_swir_b = normalize(band_green_b, band_swir_b)
band_green_f, band_swir_f = normalize(band_green_f, band_swir_f)
mndwi_b = mndwi(band_green_b, band_swir_b)
mndwi_f = mndwi(band_green_f, band_swir_f)
Anzeige der Ergebnisse mit Matplotlib
# Define a colormap that transitions from white to light green to blue to dark navy blue
colors = [(1, 1, 1), (0.8, 1, 0.8), (0, 0, 1), (0, 0, 0.4)] # (R, G, B)
cmap = LinearSegmentedColormap.from_list('custom_colormap', colors, N=256)
# Create a figure and axis for the MNDWI before flood (mndwi_b) raster
plt.figure(figsize=(8, 8))
plt.imshow(mndwi_b, cmap=cmap, vmin=-1, vmax=1) # Adjust the colormap and limits as needed
plt.colorbar(label='MNDWI')
plt.title('MNDWI Before Flood')
# Create a figure and axis for the MNDWI during flood (mndwi_f) raster
plt.figure(figsize=(8, 8))
plt.imshow(mndwi_f, cmap=cmap, vmin=-1, vmax=1) # Adjust the colormap and limits as needed
plt.colorbar(label='MNDWI')
plt.title('MNDWI During Flood')
plt.show()
Dies ist das Bild „vor der Flut“:
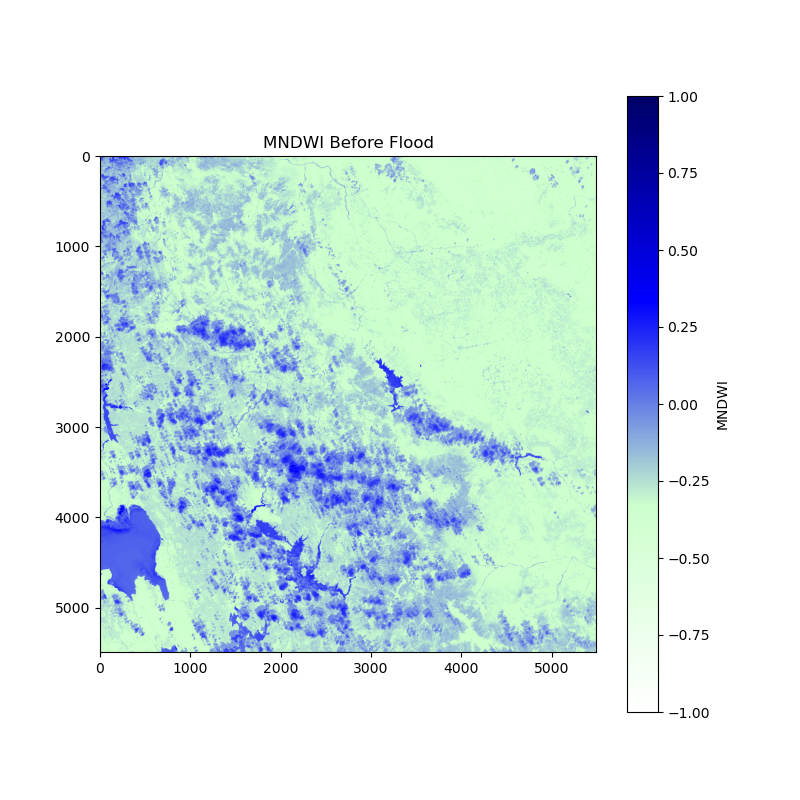
und dieses „nach der Flut“:
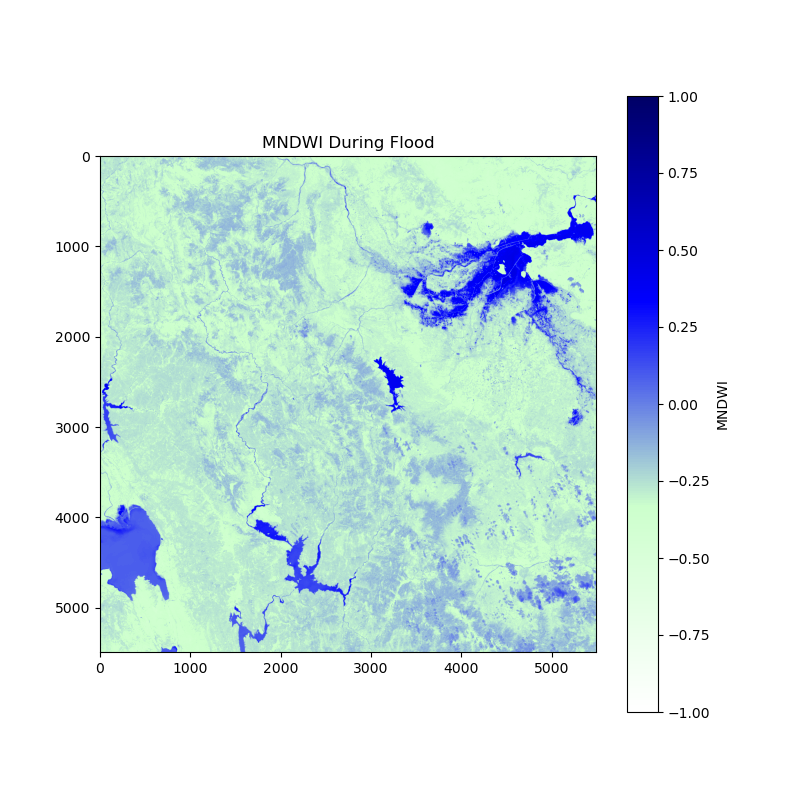
Schritt 3 Identifizierung von Gebieten mit erhöhter Wasserbedeckung
Der nächste Schritt besteht darin, Gebiete mit erhöhter Wasserpräsenz zu identifizieren. Dazu wird der MNDWI-Indexwert vor der Überschwemmung vom MNDWI-Indexwert während der Überschwemmung subtrahiert, wobei gleichzeitig die von Wolken bedeckten Gebiete ausgeschlossen werden. Das Ergebnis dieses Prozesses ist ein Bild, das die Regionen hervorhebt, in denen der Wasserstand gestiegen ist.
Ein weiteres wertvolles Produkt, das wir erhalten können, ist ein Bild, das speziell die Gebiete hervorhebt, die während des Hochwassers vom Wasser überflutet wurden. Dazu verwenden wir dieselbe Berechnung wie im vorherigen Schritt, mit einer zusätzlichen Maske, um Gebiete auszuschließen, die vor dem Hochwasser einen hohen MNDWI-Index hatten. Auf diese Weise entsteht ein Bild, das nur überflutete Gebiete zeigt.
#Create mask by SCL values
mask_f = np.logical_or(scl_f == 8, np.logical_or(scl_f == 9, scl_f == 3))
mask_b = np.logical_or(scl_b == 8, np.logical_or(scl_b == 9, scl_b == 3))
#Calculate difference between flood period and normal period
diff = mndwi_f - mndwi_b
#Calculate water tides by showing the pixcels that have positive difference values
tide = diff
tide[(diff < 0)] = 0
tide[mask_f | mask_b] = 0
tide = tide / tide.max()
#Calculate flood areas by showing the pixcels that have possitive difference values and had negative values of MNDWI before flood
flood = diff
flood[(diff < 0) | (mndwi_b > 0)] = 0
flood[mask_f | mask_b] = 0
flood = flood / flood.max()
Anzeige der Ergebnisse mit Matplotlib
# Define a colormap that goes from yellow to red with transparency for 0 values and violet for high values
colors = [(1, 1, 0, 0), (1, 0.5, 0, 1), (1, 0, 0, 1), (0.5, 0, 0.5, 1)] # (R, G, B, Alpha)
cmap = LinearSegmentedColormap.from_list('custom_colormap', colors, N=256)
# Create a figure and axis for the Water Tides raster
plt.figure(figsize=(8, 8))
plt.imshow(tide, cmap=cmap, vmin=0, vmax=np.max(tide)) # Adjust the colormap and limits as needed
plt.colorbar(label='Tide')
plt.title('Water Tides')
# Create a figure and axis for the Flood Areas raster
plt.figure(figsize=(8, 8))
plt.imshow(flood, cmap=cmap, vmin=0, vmax=np.max(flood)) # Adjust the colormap and limits as needed
plt.colorbar(label='Flood')
plt.title('Flood Areas')
plt.show()
Bild des aktuellen Wasserstandes:
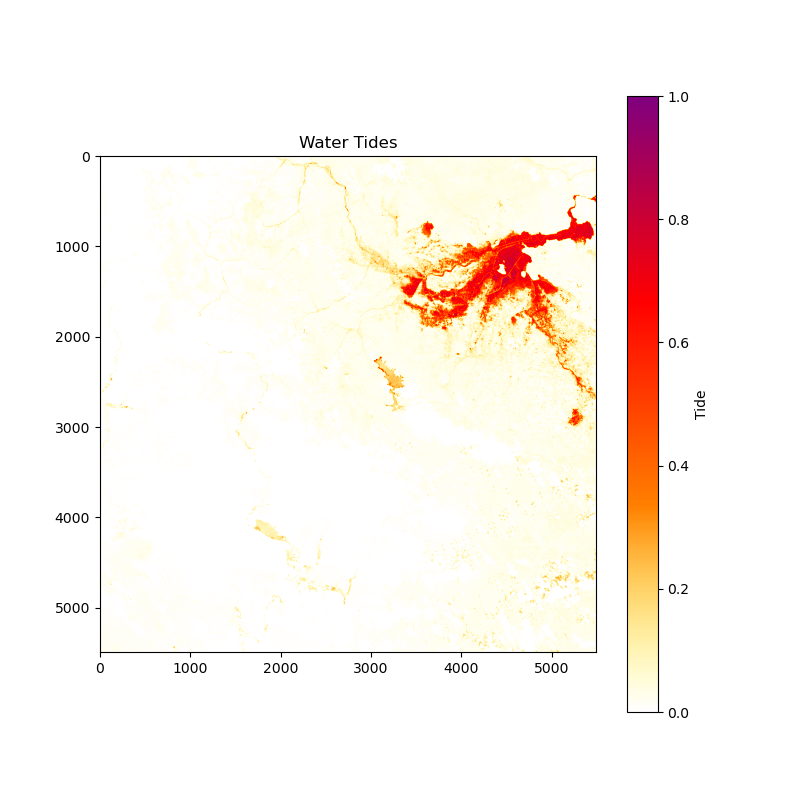
Image for Flood Area:
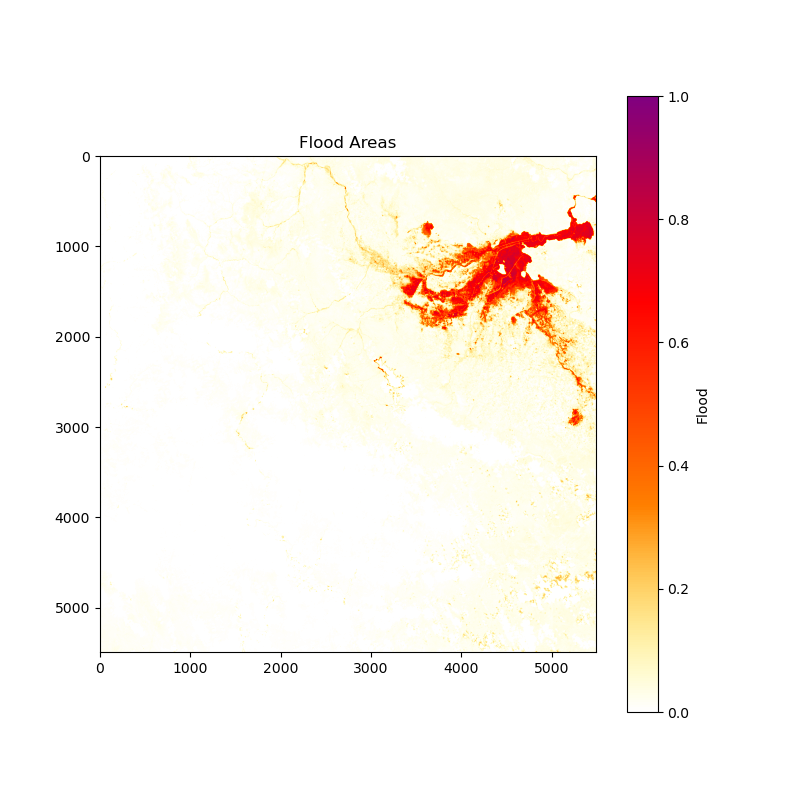
Download the resulting images
The last step will be downloading the resulting images using the Rasterio library:
def save(raster, name):
with rasterio.open(f"{name}.tif", 'w', driver='GTiff', width=raster.shape[1], height=raster.shape[0], count=1, dtype=raster.dtype, crs=crs, transform=transform) as dst:
dst.write(raster, 1)
save(mndwi_b, 'mndwi_before_greece')
save(mndwi_f, 'mndwi_flood_greece')
save(tide, 'tide_greece')
save(flood, 'flood_greece')
What To Do Next
The following article uses similar techniques for processing Sentinel-5P data on air pollution:
Verarbeitung von Sentinel-5P-Daten zur Luftverschmutzung mit Jupyter Notebook auf EO-Lab.